Von der DNA zur Protein-Primärstruktur
In diesem Projekt erstellst du ein Programm, dass eine DNA-Sequenz entgegennimmt und die resultierende Aminosäuren-Kette (Primärstruktur des Proteins) ausgibt.

Die Desoxyribonukleinsäure (DNA) ist ein langes, spiralförmiges Molekül, das die genetischen Instruktionen enthält, die notwendig sind, um ein Lebewesen zu entwickeln und am Leben zu erhalten. Diese genetischen Informationen sind in der Sequenz der vier chemischen Basen der DNA kodiert: Adenin (A), Cytosin (C), Guanin (G) und Thymin (T). Die Reihenfolge dieser Basen bestimmt die genetische Information, ähnlich wie Buchstaben Wörter und Sätze formen.
Die Proteinbiosynthese ist der Prozess, durch den die genetischen Informationen der DNA in funktionale Proteine umgesetzt werden. Dieser Vorgang findet in zwei Hauptphasen statt:
-
Transkription: Während der Transkription wird ein Abschnitt der DNA in RNA (Ribonukleinsäure) umgeschrieben. RNA ist eine einzelsträngige Kopie der DNA, die als Boten-RNA (mRNA) aus dem Zellkern in das Cytoplasma transportiert wird.
-
Translation: In der Translation liest ein Ribosom die mRNA und übersetzt die kodierten Informationen in eine spezifische Abfolge von Aminosäuren, die ein Protein bilden. Die Aminosäuren werden durch Basentripletts, die Codons, codiert.
Aminosäuren-Symbole
Es gibt 20 verschiedene Aminosäuren, die in der Natur vorkommen, und jede hat spezifische chemische Eigenschaften, die die Struktur und Funktion des Proteins beeinflussen. Hier betrachten wir aber nicht die chemischen Strukturen der Aminosäuren, sondern konzentrieren uns darauf, sie durch einfache grafische Symbole zu repräsentieren. Diese Symbole sollen helfen, die Aminosäuren visuell voneinander zu unterscheiden und ihre Präsenz in einer Aminosäurekette leicht erkennbar zu machen.
Schreibe eine Funktion aminosaeuren_kette, welche die bereits gegebene as_symbole_liste entgegennimmt und deren 4 * 5 Symbole zu einer Kette aneinandergereiht zurückgibt. Teste deine Funktion, indem du die Kette ausgibst.
Vom Codon zur Aminosäure
Da es vier verschiedene Basen gibt (Adenin, Thymin, Cytosin und Guanin in der DNA; Uracil ersetzt Thymin in der RNA), gibt es insgesamt 4^3 = 64 mögliche Codons.
Diese 64 Codons codieren für die 20 Aminosäuren, die in Proteinen vorkommen, sowie für Signale, die den Beginn und das Ende der Proteinbiosynthese anzeigen (Start- und Stopp-Codons). Die Tatsache, dass es mehr Codons als Aminosäuren gibt, führt zu einem Phänomen, das als Redundanz des genetischen Codes oder Degeneration bezeichnet wird. Das bedeutet, dass die meisten Aminosäuren durch mehr als ein Codon codiert werden - wie in der "Codesonne" unten dargestellt. Diese Redundanz hilft, die Auswirkungen von Mutationen zu minimieren, da nicht jede Veränderung in der DNA-Sequenz zu einer Änderung in der Proteinstruktur führt.
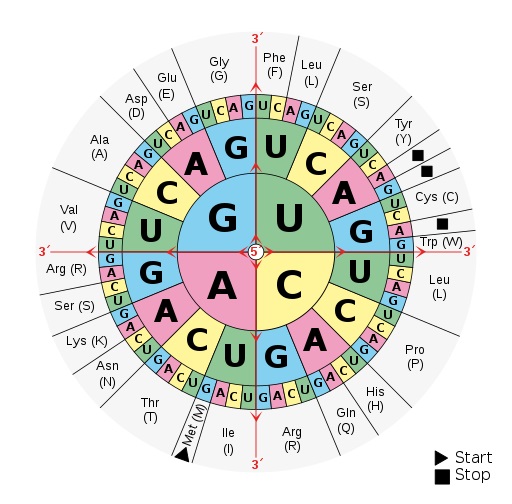
Die folgende Codezelle zeigt eine Datenstruktur, die du im Curriculum bisher nicht angetroffen hast: ein Dictionary. Wie der Name erwarten lässt und du unten sehen kannst, werden in einem Dictionary Paare bestehend aus einem Schlüssel und einem Wert (sogenannte Schlüssel-Wert-Paare) gespeichert. Dies zeigt auch die folgende Code-Schablone:
{
<Schlüssel 1> : <Wert 1>,
<Schlüssel 2> : <Wert 2>,
...
<Schlüssel n> : <Wert n>
}Beachte dabei die Verwendung von geschweiften Klammern {}.
Das Dictionary codon_as_woerterbuch listet alle möglichen Codons einer mRNA zusammen mit der jeweils codierten Aminosäure (Symbol + Beschriftung) auf. Leider ist das codon_as_woerterbuch noch lückenhaft. Nutze die "Codesonne", um die Lücken im Dictionary-Code passend zu füllen, und die Ausgabe mittels zeige_grafik zur Selbstkontrolle!
Doch wie kommt man überhaupt von der Basensequenz der DNA zu derjenigen der mRNA, und damit zu den Codons, wie sie oben im Dictionary aufgeführt sind?
Wie du bereits gelesen hast, geschieht dies durch den Prozess der Transkription. Kurz gefasst wird dabei ein bestimmter DNA-Abschnitt in eine komplementäre RNA-Sequenz umgeschrieben. Dabei wird für ein Adenin in der DNA ein Uracil in die RNA eingebaut. Weiter "paart" sich Cytosin mit Guanin, Guanin mit Cytosin und Thymin mit Adenin. Konkret wird so beispielsweise die DNA-Sequenz AGGTCTCCTAA zur mRNA-Sequenz UCCAGAGGAUU transkribiert.
Die so gebildete mRNA verlässt dann den Zellkern, um für die Proteinbiosynthese verwendet zu werden.
Entsprechend musst auch du nun eine Funktion bereitstellen, die eine gewünschte DNA-Sequenz in die entsprechende mRNA umschreibt.
Transkription
Schreibe eine Funktion transkribiere, die eine DNA-Sequenz als Parameter erwartet und die dazu passende RNA-Sequenz zurückgibt.
Translation
Nun bist du fast am Ziel!
Es fehlt noch das Übersetzen der mRNA-Informationen in die entsprechende Aminosäuren-Abfolge, auch Primärstruktur (eines Proteins) genannt. Hierbei ist zu beachten, dass die Translation erst beim sogenannten Start-Codon AUG beginnt. Damit sind auch die folgenden Codons definiert - man spricht auch vom Leseraster - und die Translation läuft solange, bis eines der Stopp-Codons UAA, UAG bzw. UGA gelesen wird.
Das Gerüst der Funktion translatiere ist bereits vorgegeben. Komplettiere die Funktion, indem du die eingefügten Kommentare sorgfältig studierst und dann in den entsprechenden Code umsetzt.
This activity has been created by Lichtsteiner and is licensed under CC BY-SA 4.0.
Von der DNA zur Protein-Primärstruktur


PyTamaro is a project created by the Lugano Computing Education Research Lab at the Software Institute of USI
Privacy Policy • Platform Version 50af0ba5 (Mon, 23 Feb 2026 11:44:41 GMT)
